|
Hybridation génomique comparativeL'hybridation génomique comparative (en anglais, Comparative Genomic Hybridization ou CGH) est une technique de cytogénétique moléculaire permettant d'analyser les variations du nombre de copies dans l'ADN. PrincipeDans un organisme diploïde tel que l'humain, chaque segment d'ADN peut être trouvé en duplicat : une copie est présente sur chacun des deux chromosomes d'une paire. Dans certaines pathologies, le nombre de copies peut varier : il peut augmenter par exemple en cas de duplication et diminuer dans le cas de délétion. L'hybridation génomique comparative permet de diagnostiquer ces pathologies et d'identifier les régions chromosomiques impliquées. 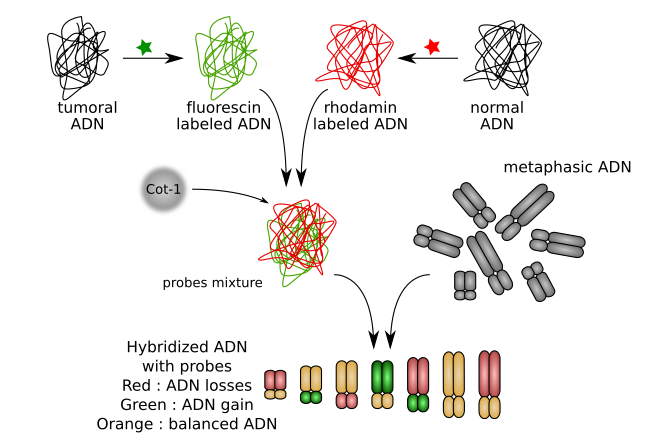 Méthode
L'ADN d'un patient est extrait de tissu normal (souvent des lymphocytes) et de tissu potentiellement tumoral puis marqué par des fluorochromes. En général, l'ADN tumoral est marqué en vert par la fluorescéine (sous forme de dUTP-FITC) et l'ADN de tissu sain en rouge par la rhodamine (sous forme de dUTP-TR). Les sondes sont mélangées ensuite avec de l'ADN Cot-1, qui permet de supprimer les séquences répétitives d'ADN, et une polymérase, qui permet de produire des fragments de 0,6 à 3 kb (milliers de paires de bases).
 Le mélange d'ADN marqué est mélangé avec de l'ADN de contrôle en métaphase. L'ADN marqué va alors s'hybrider de façon compétitive avec l'ADN métaphasique :
La fluorescence est ensuite détectée par un microscope à épifluorescence et quantifié par analyse d'image quantitative. Cette technique est capable de détecter des pertes ou des gains du nombre de copie d'ADN au niveau des chromosomes. En 2006, les référentiels indiquaient pour la sensibilité qu'il fallait des régions de 5 à 10 Mb (millions de paires de bases) pour détecter la perte d'une seule copie. En revanche, la détection d'une amplification était possible à partir de 1 Mb. Considérations techniques et limitations
Applications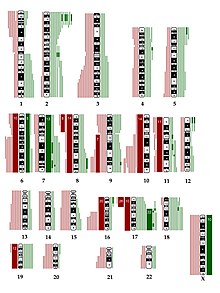
Voir aussi
Liens externes
|